Herramientas de usuario
Barra lateral
Tabla de Contenidos
EXPLORACIÓN RECURSO EDUCATIVO ABIERTO
Guía de Exploración REA 13 de mayo de 2014
En la Sesión de clase:
Como primer paso se escogió la herramienta de rasMol 2.7.5.2, el cual es de fácil descarga, de distribución gratuita; es un software utilizado para la creación de modelos moleculares para la enseñanza de la geometría molecular y espacial específicamente para química orgánica.
Descarga:
Este software se puede descargar de la página www.RasMol.org y www.OpenRasMol.org. En la parte superior de la página mencionada anteriormente, se encuentran los links con la información correspondiente a copia y seguridad, contenido, distribución, manual, historia, entre otros. Al lado izquierdo de la pantalla se selecciona el vínculo: RasMol Latest Windows Installer , al seguir las instrucciones, se evidencia que la descarga se hace en un minuto, se escoge el idioma de preferencia. Una vez instalado, se hace ingreso al programa.
RasMol es amplia gama de arquitecturas y sistemas operativos, incluyendo Microsoft Windows, Apple Macintosh, UNIX y sistemas VMS. Versiones UNIX y VMS requieren un 8, 24 o 32 bits de color de pantalla de X Window (X11R4 o posterior).
Inicio del Programa
Para empezar RasMol, se hace doble clic en el icono RasMol del gestor de programas. Al iniciar, el programa muestra una única ventana principal (la ventana de la pantalla) con un fondo negro en la pantalla y proporciona la ventana de línea de comandos, minimizada, como un pequeño icono en la parte inferior de la pantalla.
La línea de comandos o la ventana del terminal se pueden abrir haciendo doble clic en este icono RasMol. Es posible especificar un nombre de archivo de coordenadas o un nombre de archivo script o ambos en la línea de comandos de Windows. Un archivo de comandos se puede especificar mediante la adición de la opción '<filename>-script' para la línea de comandos. Una molécula de archivo de coordenadas puede cargarse colocando su nombre en la línea de comandos, opcionalmente precedido de una opción de formato de archivo.
Si no se da la opción de formato, el archivo de coordenadas especificado se supone que es en el PDB, CIF o formato mmCIF.Las opciones válidas son '-pdb', '-mdl', '-mol2', 'xyz', '- la alquimia', '-CHARMM', '-mopac' y '-cif' que corresponden al formato Protein Data Bank , el formato de Molecular Design Limited Mol archivo, formato de archivo de Tripos Sybyl Mol2, formato de archivo de MSC XMol XYZ, formato de archivo de Tripos Alchemy, formato de archivo CHARMm, formato de archivo MOPAC de JP Stewart y IUCr CIF o formato de archivo mmCIF, respectivamente.
Si se especifican tanto un archivo de coordenadas y un archivo de secuencia de comandos en la línea de comandos, la molécula se carga primero y después los comandos del script se aplican a él. Si no se encuentra ninguno de los archivos, el programa muestra el mensaje de error “Error: File not found!” y el usuario recibe el inductor de RasMol.
También es posible especificar el tamaño de la ventana de gráficos inicial o la posición o el tamaño y la posición con las opciones '-altura nnnn',' De ancho nnnn','-xpos nnnn'y'-ypos nnnn». Los valores numéricos se expresan en píxeles. La posición se especifica en términos de la esquina superior izquierda del área de representación.
La exploración se hizo seleccionando un archivo de la base de datos que se encuentra en
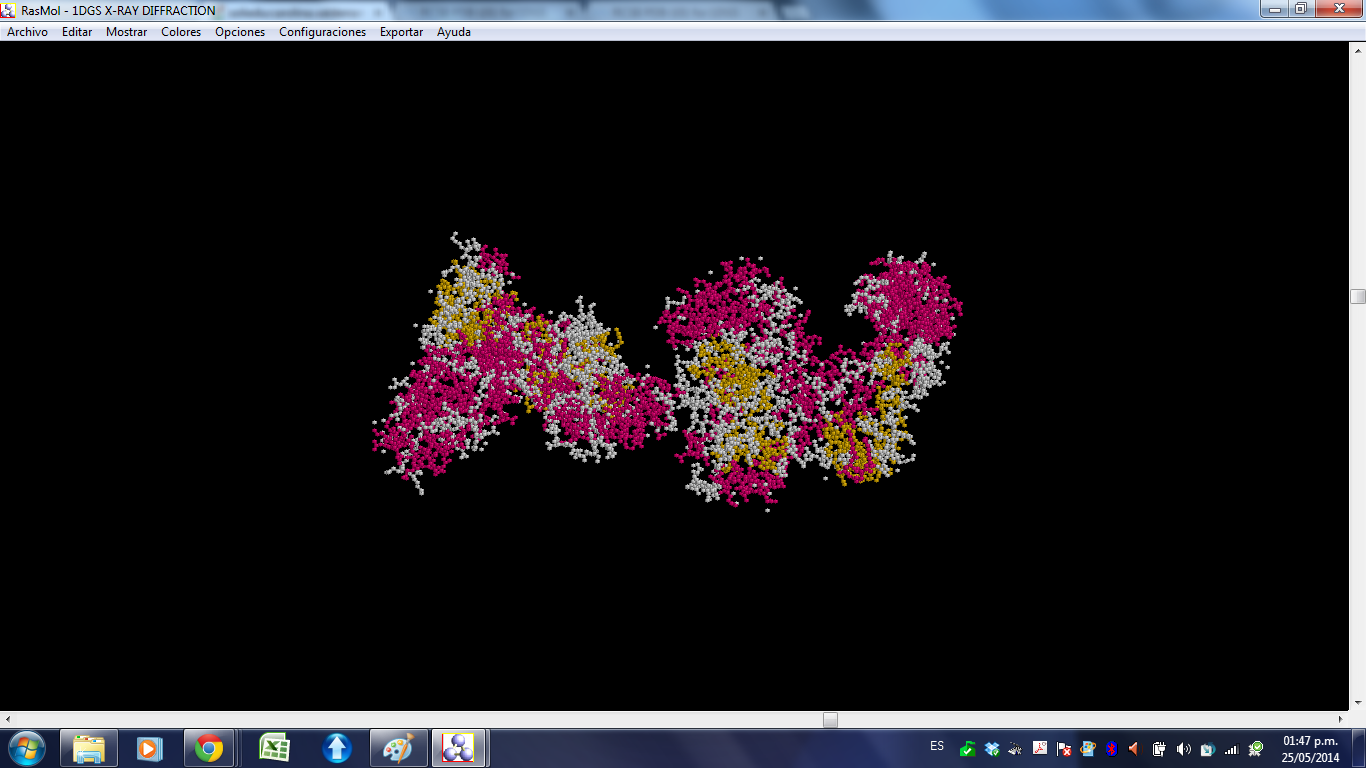
Banco de proteínas; la molécula corresponde al DNA ligase, se descargo como 1DGS.pdb, se abrió el archivo en la pantalla negra haciendo clic en el menú archivo, abrir (se busca el archivo donde se haya guardado, se selecciona) esta se visualizo en la pantalla negra, en donde se le puede hacer la manipulación tridimensional y mostrar como bastones, bolas y pines, alambre, entre otros, la pantalla de comando dice toda la información correspondiente a número de átomos, hidrógenos, número de cadenas, enlaces, giros….
Se le puede asignar colores de acuerdo a las estructuras, cadena, formas, temperatura…..
En el siguiente banco se pueden descargar imágenes .pdb de forma más sencilla:
Imagenes 3D de moléculas orgánicas
Para mayor comprensión, se puede revisó el video tutorial de you tube: Ver Tutorial
Uso:
Se hace una revisión del tutorial en you tube, el cual explica como se de ingresar para empezar a visualizar y construir las biomoléculas. el programa como ya lo mencione anteriormente es un excelente visualizador de moléculas en 3 dimensiones, que además, es gratis. Alrededor de Rasmol se ha construido toda una comunidad de usuarios, que ha generado que el programa se haya desarrollado muy rápidamente. Soporta los formatos moleculares más extendidos, como pdb, mol, mdl, xyz, etc., por lo cuál es muy sencillo encontrar moléculas en las numerosas bases de datos de la red. Por otra parte, podemos crear nuestras propias moléculas, si así lo deseamos, aunque no es necesario, como indico más arriba, pues se consiguen con gran facilidad en Internet. Existen versiones para casi todos los sistemas operativos y, es relativamente fácil encontrar manuales de uso en múltiples idiomas.
MÁS INFORMACIÓN DE INTERÉS
PROGRAMAS DE MODELIZACIÓN MOLECULAR
WebLab Viewer Pro
WebLab Viever Pro es un programa que permite crear y visualizar moléculas, desde las más sencillas a las más complejas de orgánica. Soporta múltiples formatos de moléculas y, entre ellos, los formatos pdb, mdl y xyz, de los que encontramos en la red muchas bases de datos, donde podemos obtenerlos gratuitamente. Así mismo, con el propio programa podemos buscar en Internet las moléculas que necesitemos. De cada molécula presenta la posibilidad de presentar etiquetas referidas a los átomos, los enlaces, etc. Incluso podemos añadir nosotros las etiquetas que deseemos. Otra de las ventajas que presenta es que podemos colocar como objeto OLE una molécula en un documento de Word, con lo cuál, podemos crear excelentes aplicaciones y temas de orgánica. Existe una versión Lite, gratuita, que sólo nos permite visualizar moléculas, aunque no diseñarlas, cosa que no es problema por la enorme cantidad que encontramos en la red.
Bondit
Bondit nos permite trabajar sobre el concepto de enlace, ya que con él creamos las estructuras de Lewis de las moléculas que deseemos. Su manejo es extremadamente sencillo y, partiendo de un átomo, vamos añadiendo nuevos átomos, apareciéndonos los electrones de enlace, según la regla del octeto, observando como se van formando los enlaces en las moléculas.
PROGRAMAS DE DISEÑO MOLECULAR
Chemsketch
Maravilloso programa de ACD/Labs que nos soluciona ese engorroso problema de dibujar moléculas y reacciones orgánicas. Muy fácil de manejar, pues con un simple click, podemos ir construyendo desde la molécula más simple hasta la más compleja. Presenta en su interfaz un gran número de barras de herramientas para facilitarnos todo el proceso, entre las que encontramos gran cantidad de estructuras predefinidas, que nos facilitan el mismo. Pero no terminan ahí las bondades de este excelente programa, pues con un simple click, convertimos la molécula recién dibujada, en una molécula en tres dimensiones, que observamos como si de un visor molecular se tratase. Otra ventaja es que nos permite exportar las moléculas creadas en varios formatos gráficos usuales: bmp, gif, tif, pcx, etc., lo que nos da pie a incorporarlas con gran facilidad a nuestro procesador de texto.
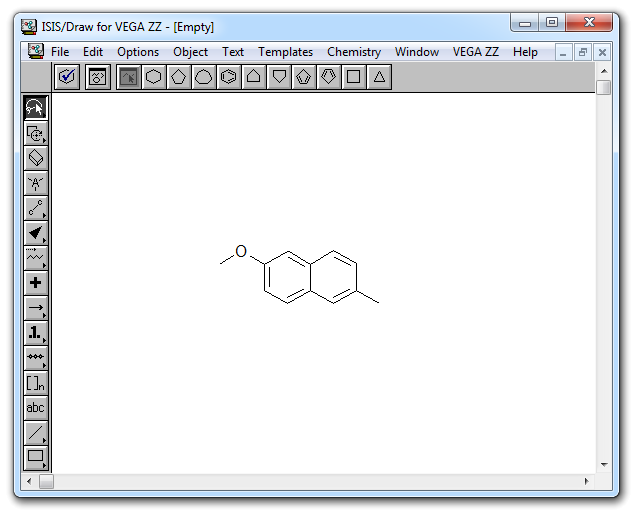
IsisDraw
Un programa de características muy similares al anterior y que presenta prácticamente sus mismas prestaciones: facilidad de uso, plantillas de moléculas prediseñadas, barras de herramientas muy completas, etc. Presenta, a diferencia del anterior programa un “inspector” que podemos ejecutar para comprobar si las reacciones que escribimos son correctas, así como una “colaboración” con el plugin CHIME, que nos permite visualizar en 2D las moléculas embebidas en las páginas web. Sin embrago no presenta la posibilidad de visualización en 3D que posee Chemsketch.
Una de sus mejores virtudes es, naturalmente, que es un programa absolutamente gratuito, aunque ello no signifique que sea un programa estático. La empresa fabricante introduce mejores con relativa asiduidad.